Mecanismos de resistencia a antibiotioticos de Mttb
Las cepas de M. tuberculosis resistentes a drogas se definen como tal por su capacidad para crecer a mayores concentraciones de antibióticos que las cepas sensibles. En otras bacterias, la resistencia genética generalmente es mediada a través de la adquisición de genes exógenos a través de plásmidos, transposones, integrones y bacteriófagos. Sin embargo, en MTB no se han reportado mecanismos de adquisición de genes de resistencia por dichos elementos. Por lo tanto, la resistencia a medicamentos anti-TB se debe principalmente a alteraciones en genes que codifican blanco de antibióticos, en productos genéticos involucrados en la activación de pro-fármacos o regiones reguladoras.
Luego existen conceptos como resistencia primaria y secundaria, la primaria o constitutiva se refiere a la infección de un paciente que nunca ha recibido tratamiento por una cepa resistente. Ésta incluye infección por cepas silvestres que nunca han estado en contacto con drogas (resistencia natural) y también la resistencia que se desarrolla como consecuencia de la exposición de una cepa a determinada droga, pero en otro paciente.
Se define como resistencia secundaria o adquirida, aquella que se desarrolla en pacientes que han recibido quimioterapia antituberculosa, debido a la selección de cepas mutantes resistentes espontáneas, en la mayoría de los casos, motivado a un tratamiento inadecuado o incumplimiento de la terapia.
Fármaco-resistencia constitutiva
La resistencia constitutiva o intrínseca, se define como la fármaco-resistencia de cualquier especie bacteriana que no ha sido adquirida como resultado de la exposición a antibióticos
Permeabilidad reducida de la pared celular mico-bacteriana

La resistencia intrínseca en MTB se ha atribuido al hecho de poseer una pared celular compleja constituida por una gran cantidad de lípidos, proteínas y polisacáridos que le confieren una característica hidrófoba con permeabilidad celular restringida para un gran número de compuestos antibacterianos. Las capas de péptidoglicano y arabinogalactano limitan la entrada de moléculas hidrófobas, mientras que la capa de ácidos micólicos limita el acceso de ambas moléculas. Antibióticos hidrofóbicos pueden ingresar a la célula por difusión a través de la bica-pa hidrofóbica, mientras que los antibióticos hidrofílicos que no se pueden difundir a través de la pared celular utilizan canales como las porinas. Hasta la fecha, las porinas MspA y OmpATb han sido identificadas y caracterizadas en m. smegmatis y MTB, respectivamente, aunque no es claro cuál es el papel que desempeñan estas proteínas en la fármaco-resistencia de micobacterias, específicamente en MTB.
Modificación y degradación enzimática de medicamentos
La producción de enzimas es quizá un mecanismo de respuesta bacteriana a los antibióticos, ya que estas utilizan una serie de estrategias para superar las propiedades tóxicas de los antibióticos. Un ejemplo de esto, es la modificación de medicamentos a través de kinasas, acetiltransferasas, adenilitransferasas, glicosiltransferasas y ADP-ribosiltransferasas. En MTB, la modificación enzimática de medicamentos se debe principalmente a la metilación del ARNr. Por ejemplo, la resistencia intrínseca de MTB a los macrólidos y lincosamidas, se debe al gen erm37, el cual codifica una metiltransferasa ARNr. Esta enzima altera las estructuras de los ribosomas de MTB a través de la metilación del 23S ARNr reduciendo la afinidad de los macrólidos y lincosamidas a los ribosomas e impidiendo su actividad inhibitoria en la síntesis de proteínas . Otro ejemplo es la metilación del ARNr que genera resistencia a capreomicina (CAP) y viomicina, dos medicamentos utilizados para tratar la TB-MDR. La CAP interactúa con las hélices 69 y 44 de las proteínas ribosomales 16S y 23S RNAr respectivamente, resultando en la inhibición de la síntesis de proteínas. Estudios genéticos, han sugerido que la resistencia a estos dos medicamentos se debe a mutaciones en el gen tlyA, el cual codifica para una metiltransferasa (2'- O-metiltransferasa) 32. El gen tlyA metila los nucleótidos C1409 en la hélice 44 de la proteína 16S ARNr y C1920 en la hélice 69 de la proteína 23S ARNr. La pérdida de estas metilaciones confiere resistencia a CAP y viomicina 33 La degradación enzimática de medicamentos, es un mecanismo que se ha estudiado principalmente en los β-lactámicos. Las β-lactamasas, hidrolizan el anillo β-lactámico proporcionando resistencia a este grupo de medicamentos. La β-lactamasa más importante en MTB es la BlaC, la cual tiene actividad contra los carbapenémicos, que son generalmente resistentes a las β-lactamasas de otras bacterias. Adicionalmente, MTB codifica otros tres genes con función β-lactamasa: blaS, rv0406c y rv3677c, los cuales han mostrado menor actividad que BlaC.
Bombas de eflujo como mecanismo de resistencia a medicamentos anti-TB
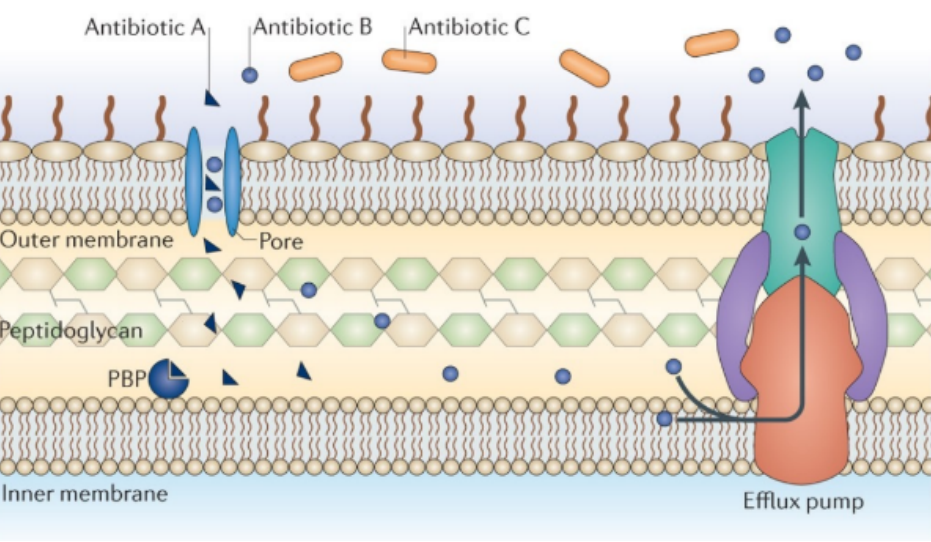
Las bombas de eflujo, son proteínas transportadoras localizadas en la membrana citoplasmática de todos los tipos de células y actúan como transportadores activos, ya que requieren una fuente de energía para realizar su función. En sistemas de eflujo bacterianos, probablemente su papel fisiológico, es proteger a la célula contra moléculas tóxicas, incluyendo antibióticos clínicamente importantes, al transportarlos una vez que ingresan a la célula bacteriana hacia el ambiente extracelular. Esta característica reduce la concentración intracelular de los antibióticos a niveles sub-1NHibitorios y de esta manera se piensa que promueven la emergencia de fármaco-resistencia, por lo que se ha sugerido que dichos sistemas confieren resistencia de bajo nivel a múltiples medicamentos, sus actividades brindan una mejor tolerancia a los medicamentos y potencian la adquisición y acumulación de mutaciones cromosomales que confieren altos niveles de resistencia. Se ha observado que de los aislamientos de MTB resistentes a medicamentos anti-TB, entre un 20-30% de los resistentes a INH, aproximadamente el 5% con resistencia a RIF y alrededor de un 15-58% de aquellos resistentes a FQS no presentan alteraciones genéticas asociadas con dicha fármaco-resistencia, lo que sugiere que otros mecanismos pueden contribuir a la fármaco-resistencia en MTB, dentro de los cuales se incluyen las bombas de eflujo.
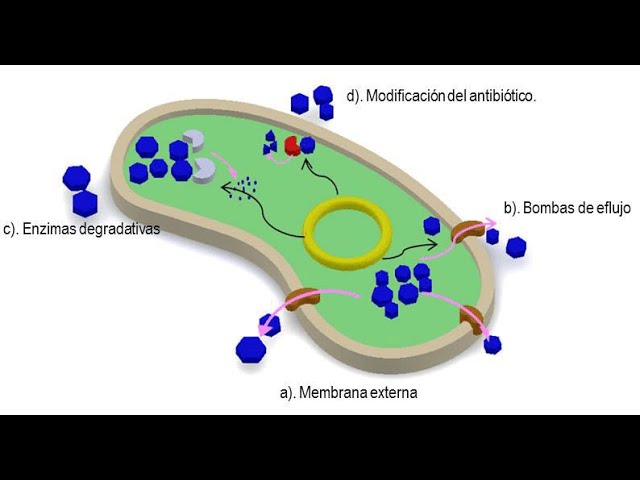
Resumen de las resistencias constitutivas
Fármaco-resistencia adquirida
Como se dijo anteriormente, en otras bacterias la resistencia genética generalmente es mediada a través de la adquisición de genes exógenos a través de plásmidos, transposones, integrones y bacteriófagos. Sin embargo, en MTB no se han reportado mecanismos de adquisición de genes de resistencia por dichos elementos. Por lo tanto, la resistencia a medicamentos anti-TB se debe principalmente a alteraciones en genes que codifican blanco de antibióticos.
Resistencia a Isoniazida (INH)

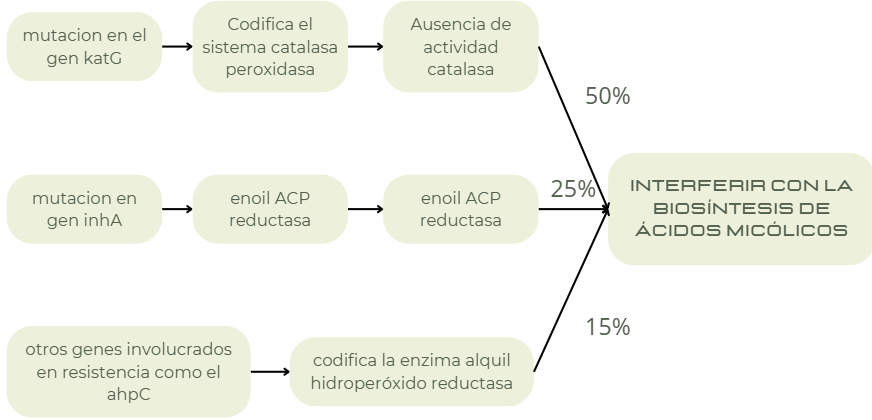
La isoniazida (ácido nicotínico hidrazida o INH) tiene acción bactericida al interferir con la biosíntesis de ácidos micólicos. Es una prodroga que al ser captada por el bacilo, es activada por el sistema catalasa-peroxidasa, de manera que la ausencia de actividad catalasa, debido a mutaciones en el gen katG, codificante de esta enzima, es uno de los mecanismos de resistencia a INH. Las cepas de M. tuberculosis con mutaciones en el gen katG exhiben poca o ninguna actividad catalasa y son altamente resistentes a INH (MIC >32 mg/ml). Las mutaciones se concentran en una región codificante del gen katG, que comprende los codones 300 al 507, siendo las más frecuentes las sustituciones de la serina 315 por treonina y el residuo de arginina 463 por leucina. Estas mutaciones explican aproximadamente el 50% de los casos de aislados clínicos resistentes a INH.
Por otra parte, la enzima enoil ACP reductasa, involucrada en los pasos de elongación de ácidos grasos, codificada por gen inhA se identificó como un blanco de acción de INH. El intermediario de INH, cuya activación depende de la actividad catalasa-peroxidasa intacta, inhibe la actividad de la enzima inhA y en consecuencia la síntesis de ácidos micólicos. Las mutaciones en el gen inhA inducen sobreexpresión del gen inhA y niveles elevados de la enzima enoil reductasa en cantidades que superan el poder inhibitorio de INH. Las mutaciones en inhA están asociadas a aproximadamente al 25% de los casos de resistencia a INH, generalmente con bajos niveles de resistencia (MIC 1mg/ml).
La investigación de otros genes involucrados en resistencia a INH que explicaran el mecanismo de resistencia del 10-20% de cepas que carecían mutaciones en katG o inhA condujo a la identificación del gen ahpC, codificante de la enzima alquil hidroperóxido reductasa, involucrada en la respuesta a estrés oxidativo. Las mutaciones en aphC están asociadas a aproximadamente un 10 a 15% de aislados clínicos resistentes a INH y actualmente se investigan otros genes candidatos asociados con resistencia a este antibiótico
Resistencia a Rifampicina (RIF)
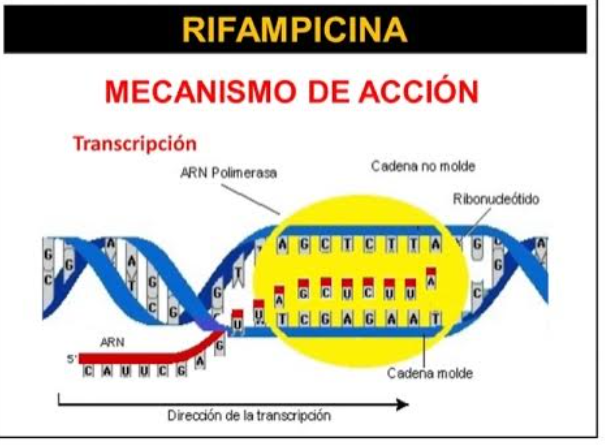
La rifampicina es un antibiótico de amplio espectro clave en la estrategia DOTS propuesta por la OMS y el mecanismo de resistencia a este antibiótico es uno de los primeros caracterizados a nivel molecular. El mecanismo de acción de la rifampicina consiste en que el antibiótico se une a la ARN polimerasa procariota, la enzima responsable del proceso de transcripción de genes, y al inhibir la expresión de genes, la rifampicina conduce a la muerte de la célula. La resistencia a rifampicina se explica por mutaciones en el gen rpoB, el cual codifica la subunidad b de la ARN polimerasa y las alteraciones en esta subunidad impiden que la rifampicina interactúe adecuadamente con la ARN polimerasa e inhiba la transcripción.
Se ha demostrado que la resistencia a rifampicina en M. tuberculosis se explica en un 95% a 98% por mutaciones en el gen rpoB, las cuales generalmente se localizan en un corto segmento de 81 pb que incluye los codones 507 a 533 del gen rpoB. Las mutaciones en esta región incluyen deleciones, inserciones, sustituciones, siendo las más frecuentes, las mutaciones en codones para asparagina 516, histidina 526 y serina 531, de manera que los métodos genotípicos para ensayar resistencia a rifampicina se basan en la detección de estas mutaciones, aunque se han encontrado mutaciones en otras regiones del gen, pero con menor frecuencia.
Resistencia a Pirazinamida

La pirazinamida es un compuesto sintético redescubierto en la década de los 80 que ha facilitado el tratamiento antituberculoso de corta duración. Su mecanismo de acción no ha sido bien dilucidado, aunque se ha señalado la importancia de la acción de la enzima pirazinamidasa. Las cepas de micobacterias susceptibles a pirazinamida sintetizan pirazinamidasa, una enzima que transforma la pirazinamida en su metabolito activo, el ácido pirazinoico, el cual además de su actividad específica parece tener la capacidad de disminuir el pH del medio intracelular por debajo de los límites de tolerancia de la bacteria. En cepas de M. tuberculosis con resistencia adquirida y M. bovis con resistencia constitutiva a pirazinamida, se han identificado interrupciones en el gen pncA, codificante de la enzima pirazinamidasa/nicotinamidasa. Entonces, uno de los mecanismos de resistencia a pirazinamida propuestos hasta el presente es la deficiencia en pirazinamidasa, con la subsecuente pérdida de la capacidad de activar el antibiótico
Resistencia a aminoglucosidos
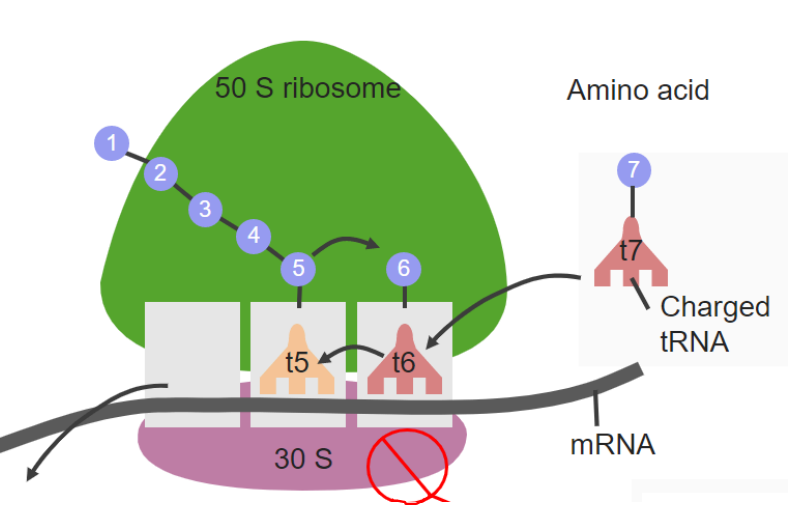
Dentro del grupo de los AMGS, la amicacina (AMK) y la kanamicina (KAN) son los medicamentos inyectables de segunda línea más usados para el tratamiento de la TB-MDR. Se ha sugerido, que la resistencia a KAN, es una de las características que define la presencia de aislamientos de MTB-XDR. El mecanismo de acción de los AMGS es INHibir la síntesis de proteínas al actuar sobre la subunidad pequeña 30S del ribosoma, específicamente en las proteínas ribosomales S12 y 16S ARNr codificadas por los genes rpsL y rrs, respectivamente. La unión al ribosoma interfiere con la elongación de la cadena peptídica causando lecturas incorrectas del código genético generando proteínas anómalas.
La estreptomicina es un antibiótico aminoglucósido bactericida que actúa sobre los ribosomas inhibiendo la síntesis de proteínas. En la mayoría de cepas de M. tuberculosis resistentes a estreptomicina se han encontrado mutaciones en el gen rpsl que codifica la proteína ribosomal S12. Otra mutación identificada, pero de menor frecuencia se localiza en el gen rrs que codifica el ARN ribosomal 16S en una región que interactúa con la proteína S12.
La resistencia fenotípica de alto nivel a los AMGS (MIC > 80 (g/ml) se ha asociado principalmente a mutaciones en la región de 1.400-bp del rrs. La mutación A1401G es la alteración más reportada, se ha identificado entre el 3090% de los aislamientos de MTB resistentes a KAN y se ha asociado con resistencia cruzada a la AMK y CAP
Resistencia a fluoroquinolonas
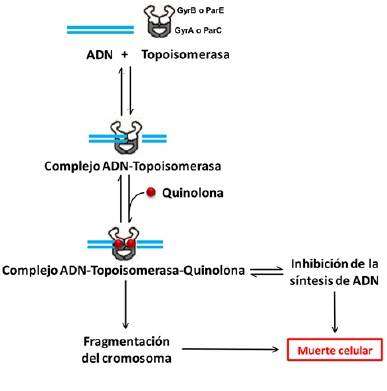
La buena distribución tisular y celular, así como los bajos efectos adversos hacen que las FQS sean utilizadas para el tratamiento de aislamientos de MTB-MDR, es usado de tratamiento empírico en comunidades con una alta tasa de MDR o el tratamiento de pacientes con reacciones adversas a los medicamentos de primera línea.
Las FQS tienen como blanco las topoisomerasas bacterianas II y IV. En las células, su función específica es catalizar la relajación y el súper enrollamiento del ADN. MTB carece de la topoisomerasa IV e incluye sólo la topoisomerasa II o ADN girasa, un tetrámero que consiste en dos subunidades A y dos subunidades B, codificadas por los genes gyrA y gyrB respectivamente
Resistencia a Etambutol
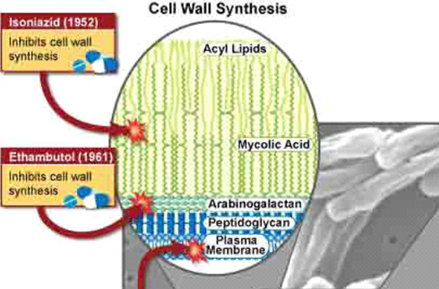
El etambutol es un compuesto sintético que actúa como bacteriostático, cuyo mecanismo de acción es inhibir la síntesis de componentes de la pared micobacteriana a las dosis habituales. Las alteraciones génicas identificadas hasta ahora se concentran en una región designada como embCAB, que incluye genes codificantes para arabinosiltransferasas (Tabla), enzimas que participan en la síntesis de componentes únicos de la pared celular de micobacterias. Las mutaciones en la región emb se asocian a altos niveles de resistencia y se han identificado en aproximadamente el 65% de los aislados clínicos resistentes a etambutol
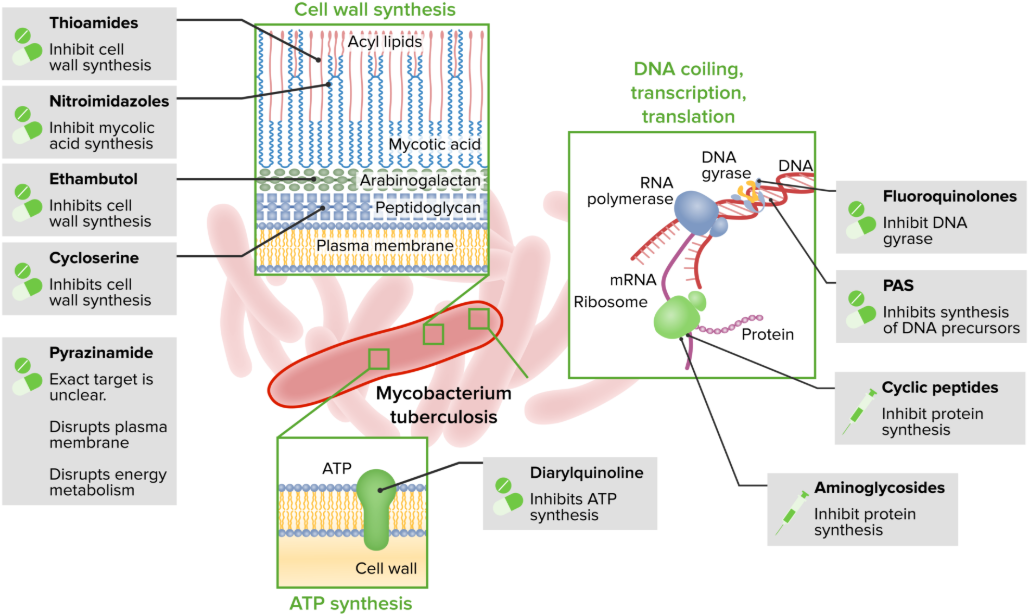
Resumen de la resistencias de mtb a los antibióticos
Clasificación según el grado de resistencia a fármacos
1. Tuberculosis multirresistente (TB-MDR)
La TB-MDR se define como la infección causada por cepas de MTB que son resistentes al menos a isoniazida (INH) y rifampicina (RIF), los dos medicamentos más potentes del régimen de primera línea. Esta forma de tuberculosis es más difícil de tratar y requiere medicamentos de segunda línea, que son menos efectivos, más tóxicos y requieren tratamientos más prolongados y costosos.
Causas frecuentes de TB-MDR:
- Prescripción médica inapropiada.
- Medicamentos de baja calidad.
- Falta de adherencia al tratamiento.
- Ausencia de programas nacionales eficaces de control.
- Supervisión inadecuada del personal de salud.
- Falta de guías estandarizadas.
- Diagnóstico tardío de la TB-MDR.
2. TB Extensamente Resistente (XDR-TB)
La XDR-TB es una forma más grave de MDR-TB. Además de la resistencia a isoniacida y rifampicina, las cepas XDR-TB son resistentes a cualquier fluoroquinolona y al menos a uno de los medicamentos del Grupo A de la OMS, como bedaquilina o linezolid. Esta resistencia adicional limita aún más las opciones de tratamiento.
3. TB Totalmente Resistente (TDR-TB)
La TDR-TB se refiere a cepas de M. tuberculosis resistentes a todos los medicamentos disponibles. Aunque se han reportado casos en países como India e Irán, la Organización Mundial de la Salud (OMS) no reconoce oficialmente esta categoría debido a la falta de una definición estandarizada y a las limitaciones en las pruebas de sensibilidad a los medicamentos.
El retraso en el diagnóstico y el tratamiento permite que las cepas resistentes se diseminen, agravando el problema a nivel poblacional.
Las micobacterias del complejo tuberculosis desarrollan resistencia a múltiples drogas por la acumulación de mutaciones individuales en varios genes, cada uno de los cuales es responsable de la resistencia a un antibiótico particular.
- Díaz, G., Galeano, J. E., & García, L. F. (2016). Resistencia de Mycobacterium tuberculosis a los medicamentos: mecanismos y alternativas terapéuticas. Revista de la Facultad de Salud Pública, 20(4), 526–537. https://doi.org/10.15446/rsap.V20n4.50575
- De Waard, J. H., & Robledo, J. (2005). Resistencia a medicamentos antituberculosos: mecanismos genéticos y diagnóstico molecular. Revista de la Sociedad Venezolana de Microbiología, 25(1), 4–11. https://ve.scielo.org/scielo.php?script=sci_arttext&pid=S0798-02642005000100004
- European Lung Foundation. (n.d.). Tuberculosis multirresistente (TBMR). Recuperado el 28 de abril de 2025, de https://europeanlung.org/es/information-hub/lung-conditions/tuberculosis-multirresistente-tbmr/
- World Health Organization (WHO). (n.d.). Tuberculosis totally drug-resistant (TDR-TB). Recuperado el 28 de abril de 2025, de https://www.who.int/news-room/questions-and-answers/item/tuberculosis-totally-drug-resistant-tb
- Caminero, J. A., García-Basteiro, A., & Rendon, A. (2021). Tuberculosis con resistencia a fármacos: nuevas recomendaciones de la OMS y su aplicación en la práctica clínica. Archivos de Bronconeumología, 57(11), 679–687. https://www.archbronconeumol.org/en-tuberculosis-con-resistencia-farmacos-nuevas-articulo-S0300289621000880